前列腺癌是全球男性最常见的泌尿系统肿瘤。根据WHO数据库 (GLOBOCAN) 数据显示,每年有超过120万新发病例和35.8万死亡病例。其中,40-70%的转移性去势抵抗前列腺癌 (mCRPC) 患者存在PTEN基因缺失。日前,多个全球临床试验中发现AKT抑制剂对基于肿瘤组织PTEN基因拷贝数缺失的前列腺癌患者有明显疗效。然而,如何通过无创伤的检测方法帮助前列腺癌患者鉴别出PTEN基因拷贝数缺失是前列腺癌临床诊疗的刚需。
继近期发表的JCOPrecision Oncology文章之后,慧渡医疗与天津医科大学第二附属医院等合作在国际专业医学杂志Frontiers in Oncology最新发表了新一代液态活检的技术突破,解决了通过血液来检测PTEN基因拷贝数缺失的国际科技难题。该技术有望为全球超过1100万前列腺癌患者带来精准治疗的福音。
Frontiers是一个领先的开放访问发布的开放科学平台,是世界上最大、被引用最多的出版商之一,其中肿瘤学期刊2020年的影响因子为6.244。本次论文的合作单位还包括来凯医药和立迪生物。

肿瘤组织取样的局限性以及陈旧的肿瘤组织的不及时性,成为制约PTEN拷贝数缺失作为预测性的肿瘤生物标志物在精准治疗发展中的瓶颈。在这项研究中,作者将慧渡医疗新一代液态活检cfDNA技术,用于mCRPC患者包括PTEN拷贝数缺失在内的、RB1和TP53基因缺失的检测,从基础研究到临床应用多个角度,探索基于血液的基因拷贝数缺失与患者预后的相关性。
- 通过PredicineCARE™技术分析15 名 ARPI 耐药的mCRPC患者队列(正交检测验证研究),检测血液循环肿瘤DNA (ctDNA) 和组织肿瘤DNA (tDNA) 的变异特征。该研究采用低通量全基因组测序 (LP-WGS) 技术进行验证,同时,用免疫组织化学 (IHC) 方法对组织样本加以验证。作者在患者血液ctDNA CNV特征中发现外周血液态活检结果与组织活检结果具有很好的一致性 (ctDNA>10%、tDNA>20%时,一致性高达81%)。
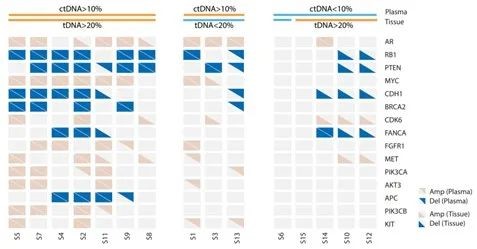
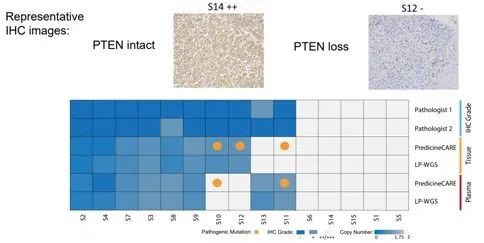
- PredicineCARE™ 给出的PTEN拷贝数缺失的检测结果,与临床金标准IHC及组织、血浆全基因组测序验证的结果高度一致。
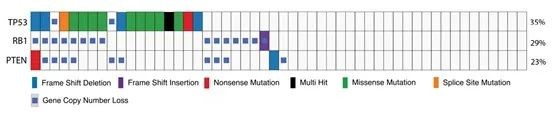
- 基于血液的 cfDNA 分析:对52例mCRPC患者血液样本的变异图谱分析发现,PredicineCARE™能精准检测出PTEN、RB1和TP53基因的7类变异类型,其中CNL变异类型在RB1和PTEN基因中占比非常高。
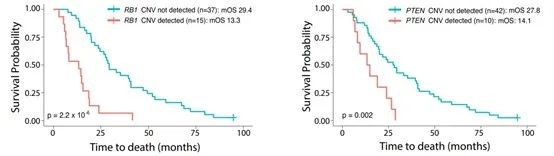
- 研究发现,存在拷贝数变异的患者总生存期 (OS) 更短 (RB1基因,mOS:29.4 VS 13.3个月;PTEN基因,mOS:27.8 VS 14.1个月)。
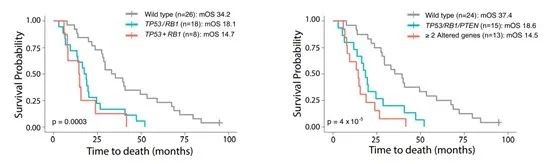
- 进一步的分析结果显示,同时检测出PTEN、RB1 和 TP53多个拷贝数缺失的mCRPC患者,相比检测出单基因CNL的患者总生存期 (OS) 更短 (TP53/RB1VS TP53+RB1, mOS: 18.1 VS 14.7个月; TP53/RB1/PTEN VS >=2个基因,mOS: 18.6 VS 14.5个月)。
作为男性泌尿系统第一高发肿瘤的前列腺癌,其中有40-70%的mCRPC患者有PTEN基因缺失。利用陈旧性肿瘤组织作为检测途径,多个全球临床试验研究发现,PTEN缺失的前列腺癌患者从AKT 抑制剂 (Afureserib, Ipatasertib, Capivasertib) 的靶向治疗中获益。慧渡医疗对血液中PTEN基因拷贝数缺失的检测,为全球上千万前列腺癌患者带来精准靶向治疗的新希望。
慧渡医疗
慧渡医疗在美国硅谷建立有完善的CLIA和CAP双认证标准实验室。通过中美两地标准一致的检测产品、统一化的实验室运营和质量体系管理,为国内外药企伙伴提供中美两地统一标准化、相互数据可比的NGS液态活检全球化、一站式精准诊断和医学大数据整体解决方案。
目前慧渡医疗已完成多项肿瘤药物治疗相关的国际临床合作研究,多篇文章发表在European Urology等影响因子大于17分的国际顶级学术期刊上。同时,慧渡医疗积极开展伴随诊断CDx全球布局,与多家全球领先的国际药企深度合作,共同开展中美伴随诊断CDx 同步开发、注册申报。
